Autoren: Sabine Hazan, Neil Stollman, Huseyin S Bozkurt, Sonya Dave, Andreas J Papoutsis, Jordan Daniels, Brad D Barrows, Eamonn MM Quigley, Thomas J Borody
Correspondence to Dr Sabine Hazan; DrHazan@progenabiome.com
Übersicht:
Was ist zu diesem Thema bereits bekannt?
Das Darmmikrobiom ist intrinsisch mit der Immunantwort des Wirts (z. B. Entzündung, Th1 vs. Th2) und der Anfälligkeit für Infektionen verbunden.
Was sind die neuen Erkenntnisse?
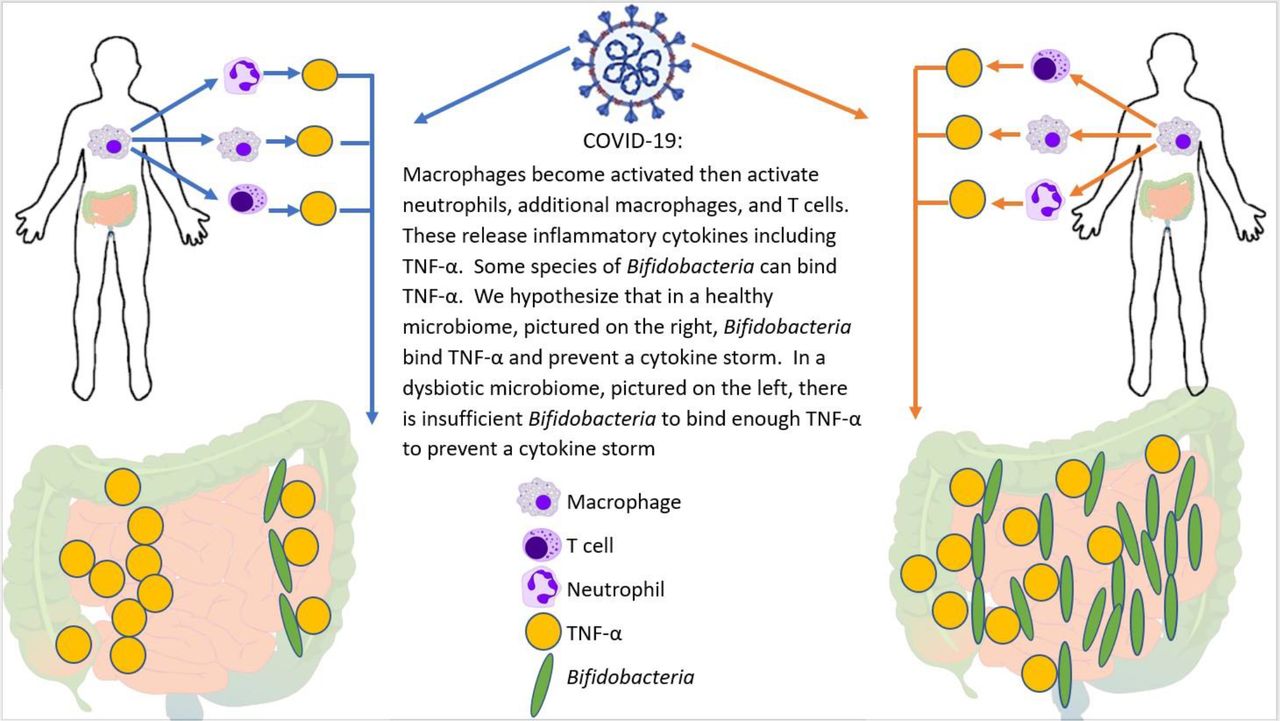
Patienten mit SARS-CoV-2-Infektionen weisen im Vergleich zu SARS-CoV-2-exponierten Kontrollen eine signifikant geringere Bakterienvielfalt, eine geringere Häufigkeit von Bifidobacterium und Faecalibacterium und eine erhöhte Häufigkeit von Bacteroides auf Gattungsebene auf. Es gibt umgekehrte Assoziationen zwischen der Schwere der Erkrankung und den Shannon- und Simpson-Diversity-Indizes sowie mit der Häufigkeit von Bifidobacterium und Faecalibacterium. Es besteht auch ein direkter Zusammenhang zwischen Schweregrad und Bacteroides-Häufigkeit.
Wie könnte es sich in absehbarer Zeit auf die klinische Praxis auswirken?
Die Verstärkung von Bifidobacterium oder Faecalibacterium durch probiotische Supplementierung oder fäkale Mikrobiotatransplantation ist es wert, bei der Behandlung von Patienten mit akuter schwerer Erkrankung oder langwieriger Infektion untersucht zu werden. Wenn die von uns dokumentierten Veränderungen bei den am stärksten Betroffenen einer SARS-CoV-2-Infektion vorausgehen, kann dieser Therapieansatz von besonderem Interesse sein. Umgekehrt, wenn die Reduktion auf eine Infektion folgt, kann die Wiederbesiedlung des Darmmikrobioms die langfristigen Auswirkungen im Zusammenhang mit Veränderungen der Zusammensetzung des Darmmikrobioms bei einer SARS-CoV-2-Infektion verringern.
Abstrakt
Ziel Ziel der Studie war es, die Diversität und Zusammensetzung des Darmmikrobioms bei SARS-CoV-2-PCR-positiven Patienten, deren Symptome von asymptomatisch bis schwer reichten, mit PCR-negativen exponierten Kontrollen zu vergleichen.
Design Unter Verwendung eines Querschnittsdesigns führten wir eine Sequenzierung der nächsten Generation mit Schrotflinte an Stuhlproben durch, um die Zusammensetzung und Diversität des Darmmikrobioms bei beiden Patienten mit SARS-CoV-2-PCR-bestätigten Infektionen zu bewerten, die sich im März bei Ventura Clinical Trials zur Behandlung vorgestellt hatten 2020 bis Oktober 2021 und SARS-CoV-2 PCR-negative exponierte Kontrollen. Die Patienten wurden basierend auf den Kriterien des National Institute of Health als asymptomatisch oder mit leichten, mittelschweren oder schweren Symptomen klassifiziert. Exponierte Kontrollen waren Personen mit längerem oder wiederholtem engem Kontakt mit Patienten mit einer SARS-CoV-2-Infektion oder ihren Proben, z. B. Haushaltsmitglieder von Patienten oder medizinisches Personal an vorderster Front. Die Diversität und Zusammensetzung des Mikrobioms wurden zwischen Patienten und exponierten Kontrollen auf allen taxonomischen Ebenen verglichen.
Ergebnisse Verglichen mit Kontrollen (n=20) hatten schwer symptomatische SARS-CoV-2-infizierte Patienten (n=28) eine signifikant geringere Bakterienvielfalt (Shannon-Index, p=0,0499; Simpson-Index, p=0,0581) und insgesamt positive Patienten hatten geringere relative Häufigkeiten von Bifidobacterium (p < 0,0001), Faecalibacterium (p = 0,0077) und Roseburium (p = 0,0327), während sie erhöhte Bacteroides (p = 0,0075) aufwiesen. Interessanterweise gab es einen umgekehrten Zusammenhang zwischen der Schwere der Erkrankung und der Häufigkeit derselben Bakterien.
Schlussfolgerung Wir gehen davon aus, dass eine geringe Bakterienvielfalt und die Verarmung von Bifidobacterium-Gattungen entweder vor oder nach der Infektion zu einer reduzierten Proimmunfunktion führten, wodurch eine SARS-CoV-2-Infektion symptomatisch werden konnte. Dieses besondere Dysbiosemuster kann ein Anfälligkeitsmarker für die symptomatische Schwere einer SARS-CoV-2-Infektion sein und kann für eine Intervention vor der Infektion, während der Infektion oder nach der Infektion zugänglich sein.
Studienregistrierungsnummer NCT04031469 (PCR−) und 04359836 (PCR+).